Sie können jeden Bibliothekspool einreichen, der mit Illumina®-Sequenzierung kompatibel ist, einschließlich solcher, die aus genomischer DNA für Whole-Genome-Sequenzierung, RNA für Transkriptomanalysen oder Amplicon-Bibliotheken hergestellt wurden. Bitte beachten Sie, dass das Pooling verschiedener Bibliotheken zu einem finalen einsatzbereiten Pool von den Kunden selbst durchgeführt werden muss – dieser Service wird von Eurofins Genomics nicht angeboten.
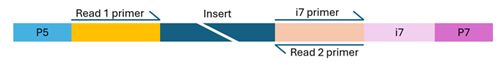
Illumina-kompatible Bibliotheken müssen vier zentrale Komponenten in ihren Adaptersequenzen enthalten – diese befinden sich an den Enden jedes Fragments – um eine erfolgreiche Paired-End-Sequenzierung zu ermöglichen:
- Flow-Cell-Bindungsstellen (P5 und P7): Ermöglichen die Bindung und Clusterbildung der DNA-Fragmente auf der Flow Cell.
- Sequenzierprimer-Bindungsstellen (Read 1 und Read 2): Starten die Sequenzierung von beiden Enden des DNA-Fragments.
- Indexsequenzen (i5 und i7): Auch als Barcodes bekannt, ermöglichen sie das Multiplexing von Proben. Bibliotheken können einfach (ein Index) oder doppelt (zwei Indizes) indiziert sein. Für NovaSeq liegt die empfohlene Barcodelänge bei 8–12 bp, für MiSeq empfehlen wir maximal 10 bp lange Indexsequenzen.
- Indexprimer-Bindungsstellen: Diese sind notwendig, um die Indexsequenzen während der Sequenzierung auszulesen.
Single-Index Bibliothek
Dual-Index Bibliothek
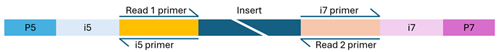
Die Struktur der Adaptersequenzen variiert je nachdem, ob die Bibliothek mit einfacher oder doppelter Indizierung arbeitet. Fehlende, falsch konfigurierte oder stark modifizierte Komponenten können dazu führen, dass die Sequenzierung fehlschlägt. Wenn Sie sich bezüglich der Konfiguration Ihrer Bibliothek unsicher sind, können Sie sich gerne an uns wenden – wir beraten Sie gerne.